לאחר שהחוקר האמריקני אריק ס. לנדר הציע רשמית פולימורפיזם נוקלאוטיד בודד (SNP) כסמן מולקולרי של הדור השלישי בשנת 1996, נעשה שימוש נרחב ב-SNP בניתוח אסוציאציות כלכליות, בניית מפת קישורים גנטית ביולוגית והקרנה של גנים פתוגניים בבני אדם., אבחון וחיזוי סיכון מחלות, בדיקת תרופות פרטנית ותחומי מחקר ביולוגיים ורפואיים אחרים.בתחום גידול יבול מזומן, זיהוי SNP יכול לממש בחירה מוקדמת של תכונות נדרשות.לבחירה זו יש מאפיינים של דיוק גבוה והיא יכולה למנוע ביעילות את הפרעות מורפולוגיה וגורמים סביבתיים, ובכך לקצר מאוד את תהליך הרבייה.לכן, ל-SNP תפקיד עצום בתחום המחקר הבסיסי.
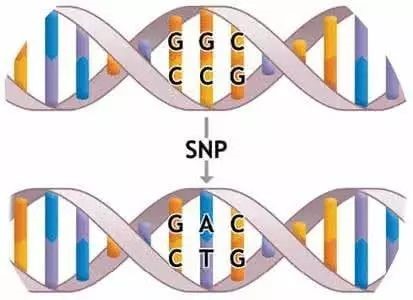
פולימורפיזם נוקלאוטיד בודד (Single Nucleotide Polymorphism, SNP) מתייחס לתופעה שיש הבדלים נוקלאוטידים בודדים באותו מיקום ברצף ה-DNA של פרטים מאותו מין או שונה.ההכנסה, המחיקה, ההמרה וההיפוך של בסיס בודד יכולים לגרום להבדל זה.בעבר, ההגדרה של SNP הייתה שונה מזו של מוטציה.לוקוס וריאנטי מחייב שתדירות אחד מהאללים באוכלוסייה תהיה גדולה מ-1% על מנת להיות מוגדר כלוקוס SNP.עם זאת, עם התרחבות התיאוריות הביולוגיות המודרניות ויישום הטכנולוגיה, תדירות אללים אינה עוד תנאי הכרחי להגבלת ההגדרה של SNP.על פי נתוני וריאציות הנוקלאוטידים הבודדות הכלולים במסד הנתונים של Single Nucleotide Polymorphisms (dbSNP) תחת המרכז הלאומי למידע ביוטכנולוגיה (NCBI), נכללים גם החדרה/מחיקה בתדר נמוך, וריאציה של מיקרו-לווין וכו'.
בגוף האדם, התדירות של SNP היא 0.1%.במילים אחרות, יש ממוצע של אתר SNP אחד לכל 1000 זוגות בסיסים.למרות שתדירות ההתרחשות גבוהה יחסית, לא כל אתרי SNP יכולים להיות סמנים מועמדים הקשורים לתכונות.זה קשור בעיקר למיקום שבו ה-SNP מתרחש.
תיאורטית, SNP יכול להתרחש בכל מקום ברצף הגנום.SNPs המתרחשים באזור המקודד יכולים לייצר מוטציות נרדפות ומוטציות לא נרדפות, כלומר, חומצת האמינו משתנה או לא משתנה לפני ואחרי המוטציה.חומצת האמינו שהשתנתה גורמת בדרך כלל לשרשרת הפפטידים לאבד את תפקידה המקורי (מוטציית missense), ועלולה גם לגרום להפסקת תרגום (מוטציית שטות).SNPs המופיעים באזורים שאינם מקודדים ובאזורים בין-גניים עשויים להשפיע על שחבור mRNA, על הרכב רצף ה-RNA הלא-מקודד ועל יעילות הקישור של גורמי שעתוק ו-DNA.הקשר הספציפי מוצג באיור:
סוגי SNP:
מספר שיטות הקלדת SNP נפוצות והשוואה ביניהן
על פי עקרונות שונים, שיטות זיהוי SNP נפוצות מחולקות לקטגוריות הבאות:
השוואת סיווג של שיטות זיהוי
הערה: הרשומות בטבלה משמשות כיום שיטות זיהוי נפוצות יותר של SNP, שיטות זיהוי אחרות כגון הכלאה של אתר ספציפי (ASH), הרחבת פריימר של אתר ספציפי (ASPE), הרחבת בסיס יחיד (SBCE), חיתוך אתר ספציפי (ASC), טכנולוגיית שבב גנים, טכנולוגיית ספקטרומטריית מסה וכו' לא סווגו והשוו.
העלות והזמן של טיהור חומצת גרעין במספר שיטות זיהוי SNP נפוצות לעיל הם בלתי נמנעים.עם זאת, ערכות קשורות המבוססות על טכנולוגיית PCR ישירה של Foregene יכולות לבצע ישירות הגברה של PCR או qPCR על דגימות לא מטוהרות, מה שמביא נוחות חסרת תקדים לזיהוי SNP.
מוצרי סדרת PCR ישירה של Foregene משמיטים בפשטות ובגסות את שלבי טיהור הדוגמאות, מה שמפחית מאוד את הזמן והעלות הנדרשים להכנת תבניות.ל-Taq פולימראז הייחודי יש יכולת הגברה מעולה והוא יכול לסבול מגוון מעכבים מסביבות הגברה מורכבות.מאפיינים אלו מספקים ערובה טכנית להשגת מוצרים ספציפיים בעלי תשואה גבוהה. ערכות PCR/qPCR של Foregene Direct לסוגי דגימות שונים, כגון: רקמות בעלי חיים (זנב חולדה, דג זברה וכו'), עלי צמחים, זרעים (כולל דגימות פוליסכרידים ופוליפנולים) וכו'.
זמן פרסום: 23 ביולי 2021