חומר מוצא: RNA
PCR של שעתוק הפוך כמותי (RT-qPCR) היא שיטה ניסיונית המשמשת בניסויי PCR תוך שימוש ב-RNA כחומר המוצא.בשיטה זו, RNA הכולל או RNA שליח (mRNA) מועתק לראשונה ל-DNA משלים (cDNA) על ידי תמלול הפוך.לאחר מכן, בוצעה תגובת qPCR תוך שימוש ב-cDNA כתבנית.נעשה שימוש ב-RT-qPCR במגוון יישומי ביולוגיה מולקולרית, כולל ניתוח ביטוי גנים, אימות הפרעות RNA, אימות מיקרו-מערך, זיהוי פתוגנים, בדיקות גנטיות וחקר מחלות.
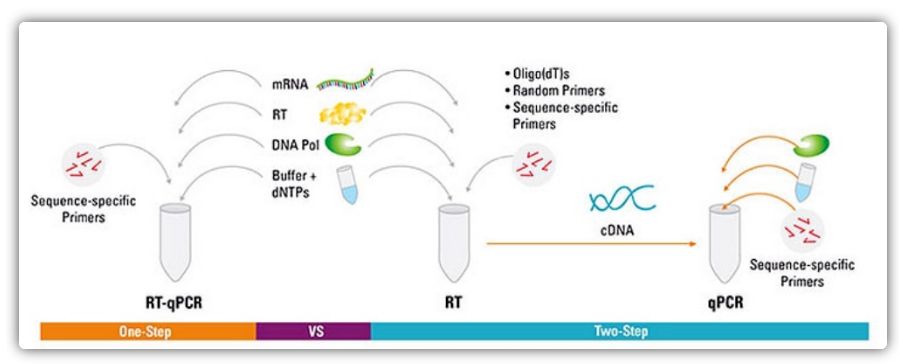
שיטות שלב אחד ודו-שלבי עבור RT-qPCR
ניתן לבצע RT-qPCR בשיטה חד-שלבית או דו-שלבית.RT-qPCR חד-שלבי משלב שעתוק הפוך והגברת PCR, מה שמאפשר ל-Reverse Transcriptase ו-DNA פולימראז להשלים את התגובה באותו צינור באותם תנאי חיץ.RT-qPCR חד-שלבי דורש רק שימוש בפריימרים ספציפיים לרצף.ב-RT-qPCR דו-שלבי, שעתוק הפוך והגברת PCR מבוצעים בשתי צינורות, תוך שימוש במאגרים אופטימליים שונים, תנאי תגובה ואסטרטגיות עיצוב פריימר.
| יתרון | חִסָרוֹן | |
| צעד אחד | לשיטה זו יש פחות שגיאות ניסיוניות שכן שתי התגובות נעשות בצינור אחד
פחות שלבי פיפטציה מפחיתים את הסיכון לזיהום
מתאים להגברה/הקרנה בתפוקה גבוהה, מהיר וניתן לשחזור | לא ניתן לבצע אופטימיזציה של תגובות דו-שלביות בנפרד
מכיוון שתנאי התגובה נפגעים על ידי שילוב התגובה הדו-שלבית, הרגישות אינה טובה כמו זו של השיטה הדו-שלבית
מספר המטרות שזוהו על ידי מדגם בודד קטן |
| שני שלבים | יכולת ליצור ספריות cDNA יציבות שניתן לאחסן לפרקי זמן ארוכים ולהשתמש בהן במספר תגובות
ניתן להגביר גנים מטרה וגני ייחוס מאותה ספריית cDNA ללא צורך במספר ספריות cDNA
מאגרי תגובה ותנאי תגובה המאפשרים אופטימיזציה של ריצות תגובה בודדות
בחירה גמישה של תנאי טריגר | שימוש במספר צינורות, ועוד שלבי פיפטציה מגבירים את הסיכון לזיהום DNA, וגוזל זמן.
דורש יותר אופטימיזציה מאשר השיטה החד-שלבית |
מוצרים קשורים:
RT-qPCR Easyᵀᴹ (שלב אחד)-SYBR ירוק I
RT-qPCR Easyᵀᴹ (צעד אחד)-Taqman
RT Easyᵀᴹ I Master Premix עבור סינתזת CDNA ברצף ראשון
זמן אמת PCR Easyᵀᴹ-SYBR Green I Kit
בחירה של סך RNA ו-mRNA
בעת תכנון ניסוי RT-qPCR, חשוב להחליט אם להשתמש ב-RNA הכולל או ב-mRNA מטוהר כתבנית לשעתוק הפוך.למרות ש-mRNA עשוי לספק רגישות מעט גבוהה יותר, עדיין נעשה שימוש תדיר ב-RNA הכולל.הסיבה לכך היא שלרנ"א הכולל יש יתרון חשוב יותר כחומר מוצא מאשר mRNA.ראשית, התהליך דורש פחות שלבי טיהור, מה שמבטיח שחזור כמותי טוב יותר של התבנית ונורמליזציה טובה יותר של התוצאות למספרי התאים ההתחלתיים.שנית, הוא נמנע משלב העשרת ה-mRNA, שיכול למנוע את האפשרות של תוצאות מוטות עקב התאוששות שונה של mRNA שונים.בסך הכל, מכיוון שברוב היישומים הכימות היחסי של גן המטרה חשוב יותר מהרגישות המוחלטת של הגילוי, RNA הכולל מתאים יותר ברוב המקרים.
פריימר לשעתוק הפוך
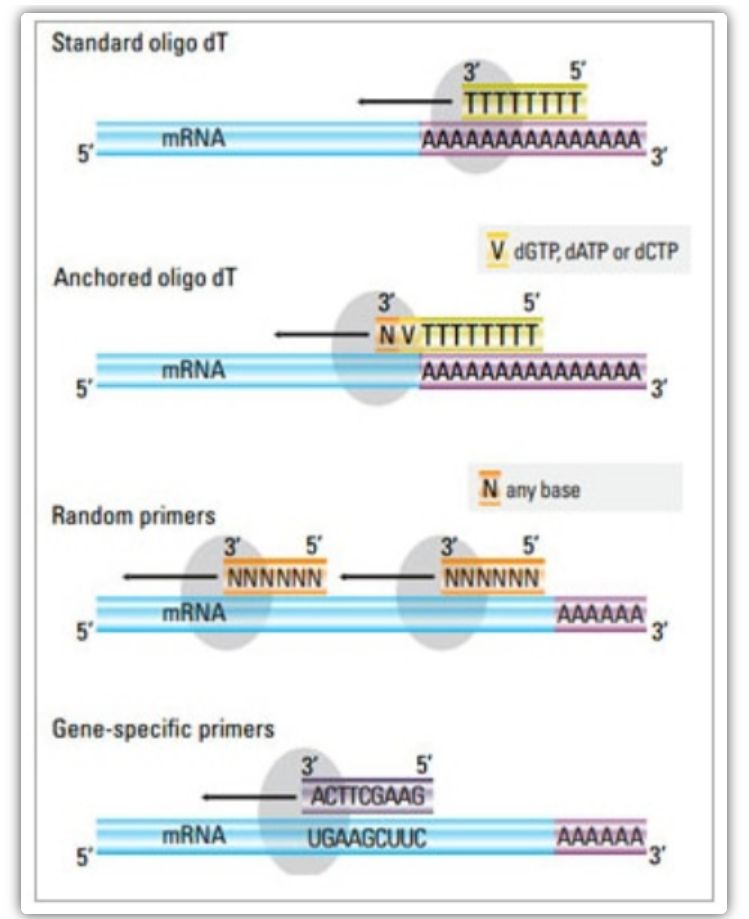
בשיטה הדו-שלבית, ניתן להשתמש בשלוש שיטות שונות כדי לקדם את תגובת ה-cDNA: פריימרים oligo(dT), פריימרים אקראיים או פריימרים ספציפיים לרצף.בדרך כלל, primers oligo(dT) ופריימרים אקראיים משמשים בשילוב.פריימרים אלו מתחלשים לגדיל ה-mRNA של התבנית ומספקים תעתיק הפוך עם נקודת מוצא לסינתזה.
| בחירת פריימר | מבנה ותפקוד | יתרון | חִסָרוֹן |
| פריימר Oligo(dT) (או פריימר אוליגו(dT) מעוגן) | חישול מורחב לשאריות תימין בזנב ה-poly(A) של mRNA;פריימר anchor oligo(dT) מכיל G, C או A בקצה 3' (אתר עוגן) | סינתזה של cDNA באורך מלא מ-mRNA בעל זנב פולי(A).
ישים כאשר פחות חומר מוצא זמין
אתר העיגון מבטיח שהפריימר אוליגו(dT) נקשר לזנב הפולי(A) 5′ של ה-mRNA | מתאים רק להגברת גנים עם זנבות פולי(A).
השג cDNA קטוע מאתר ה-priming*2 ב-poly(A)
מוטה לאגד לקצה 3′*
*אפשרות זו ממוזערת אם משתמשים בפריימרים מעוגנים של אוליגו(dT). |
| פריימר אקראי
| באורך של 6 עד 9 בסיסים, שיכולים להיצמד לאתרים מרובים במהלך שעתוק RNA | חישול לכל ה-RNA (tRNA, rRNA ו-mRNA)
מתאים לתמלילים בעלי מבנה משני משמעותי, או כאשר פחות חומר מוצא זמין
תפוקת cDNA גבוהה | cDNA מתועתק לאחור מכל ה-RNA, שבדרך כלל אינו רצוי ועלול לדלל את האות של ה-mRNA היעד
לקבל cDNA קטוע |
| פריימרים ספציפיים לרצף | פריימרים מותאמים אישית המכוונים לרצפי mRNA ספציפיים | ספריית cDNA ספציפית
שפר את הרגישות
שימוש בפריימרים הפוך של qPCR | מוגבל רק לסינתזה של גן מטרה יחיד |
תמלול הפוך
טרנסקריפטאז הפוך הוא אנזים המשתמש ב-RNA כדי לסנתז DNA.לחלק מהתעתיקים הפוכים יש פעילות RNase ויכולים לפרק גדילי RNA בגדילים היברידיים של RNA-DNA לאחר שעתוק.אם אין לו פעילות אנזימטית של RNase, ניתן להוסיף RNaseH ליעילות qPCR גבוהה יותר.אנזימים בשימוש נפוץ כוללים טרנסקריפטאז הפוך של נגיף לוקמיה של עכברים Moloney ו-reverse transcriptase של נגיף המיאלובלסטומה של העופות.עבור RT-qPCR, אידיאלי לבחור ב-reverse transcriptase עם יציבות תרמוסית גבוהה יותר, כך שניתן לבצע סינתזה של cDNA בטמפרטורות גבוהות יותר, מה שמבטיח שעתוק מוצלח של RNAs עם מבנה משני גבוה יותר, תוך שמירה על פעילותם המלאה לאורך כל התגובה, וכתוצאה מכך תשואות cDNA גבוהות יותר.
מוצרים קשורים:
פעילות RNase H של transcriptase הפוכה
RNaseH מסוגל לפרק גדילי RNA מדופלקסים של RNA-DNA, מה שמאפשר סינתזה יעילה של DNA דו-גדילי.עם זאת, כאשר משתמשים ב-mRNA ארוך כתבנית, ה-RNA עלול להתפרק בטרם עת, וכתוצאה מכך cDNA קטוע.לכן, לעתים קרובות מועיל למזער את פעילות RNaseH במהלך שיבוט cDNA אם יש צורך בסינתזה של תמלילים ארוכים.לעומת זאת, תעתיקים הפוכים עם פעילות RNase H מועילים לעתים קרובות ליישומי qPCR מכיוון שהם משפרים את ההיתוך של דופלקסים של RNA-DNA במהלך המחזור הראשון של PCR.
עיצוב פריימר
פריימרים של PCR המשמשים לשלב qPCR ב-RT-qPCR צריכים להיות מתוכננים באופן אידיאלי כדי להשתרע על צומת אקסון-אקסון, כאשר פריימר הגברה עשוי להשתרע על גבול אקסון-אינטררון בפועל.מכיוון שרצפי DNA גנומי המכילים אינטרונים אינם מוגברים, עיצוב זה מפחית את הסיכון לתוצאות חיוביות שגויות המוגברות מזיהום DNA גנומי.
אם לא ניתן לעצב פריימרים להפריד אקסונים או גבולות אקסון-אקסון, ייתכן שיהיה צורך לטפל בדגימות RNA עם DNase I או dsDNase ללא RNase כדי להסיר זיהום DNA גנומי.
בקרת RT-qPCR
יש לכלול בקרת תמלול הפוך (בקרת-RT) בכל ניסויי RT-qPCR כדי לזהות זיהום DNA (כגון DNA גנומי או תוצרי PCR מתגובות קודמות).בקרה זו מכילה את כל מרכיבי התגובה למעט טרנסקריפטאז הפוך.מאחר שתעתוק הפוך אינו מתרחש עם בקרה זו, אם נצפה הגברה של PCR, סביר להניח כי זיהום מ-DNA קיים.
זמן פרסום: אוגוסט-02-2022








